Funções, objetos e classes
Fernando P. Mayer
1 Funções e argumentos
As funções no R são definidas como:
nome(argumento1, argumento2, ...)Exemplo: função runif() (para gerar valores aleatórios
de uma distribuição uniforme):
runif(n, min = 0, max = 1)runif(10, 1, 100)# [1] 82.42955 97.51117 46.93282 21.90022 34.14275 9.44935 92.89543 19.88535
# [9] 62.39317 32.62787Argumentos que já possuem um valor especificado (como
max e min) podem ser omitidos:
runif(10)Se os argumentos forem nomeados, a ordem deles dentro da função não tem mais importância:
runif(min = 1, max = 100, n = 10)Argumentos nomeados e não nomeados podem ser utilizados, desde que os não nomeados estejam na posição correta:
runif(10, max = 100, min = 1)1.1 Outros tipos de argumentos
Exemplo: função sample():
sample(x, size, replace = FALSE, prob = NULL)xesizedevem ser obrigatoriamente especificadosreplaceé lógico:TRUE(T) ouFALSE(F)probé um argumento vazio ou ausente (“opcional”)
Exemplo: função plot():
plot(x, y, ...)- “
...” permite especificar argumentos de outras funções (por exemplopar())
Para ver todos os argumentos disponíveis de uma função, podemos usar
a função args()
args(sample)# function (x, size, replace = FALSE, prob = NULL)
# NULL2 Mecanismos de ajuda
Argumentos e detalhes do funcionamento das funções:
?runifou
help(runif)A documentação contém os campos:
- Description: breve descrição
- Usage: função e todos seus argumentos
- Arguments: lista descrevendo cada argumento
- Details: descrição detalhada
- Value: o que a função retorna
- References: bibliografia relacionada
- See Also: funções relacionadas
- Examples: exemplos práticos
Procura por nomes de funções que contenham algum termo:
apropos("mod")
apropos("model")Procura por funções que contenham palavra em qualquer
parte de sua documentação:
help.search("palavra")Ajuda através do navegador (também contém manuais, …):
help.start()Sites para busca na documentação dos diversos pacotes:
- RDocumentation https://www.rdocumentation.org/
- R Package Documentation https://rdrr.io/
- R Contributed Documentation (várias línguas) https://cran.r-project.org/other-docs.html
Os pacotes do R contém funções específicas para determinadas tarefas, e estendem a instalação básica do R. Atualmente existem mais de 10000 pacotes disponíveis no CRAN, além de diversos outros hospedados em sites como Github, por exemplo.
Ao instalar o R, os seguintes pacotes já vêm instalados (fazem parte do chamado “R core”):
# [1] "base" "boot" "class" "cluster" "codetools"
# [6] "compiler" "datasets" "foreign" "graphics" "grDevices"
# [11] "grid" "KernSmooth" "lattice" "MASS" "Matrix"
# [16] "methods" "mgcv" "nlme" "nnet" "parallel"
# [21] "rpart" "spatial" "splines" "stats" "stats4"
# [26] "survival" "tcltk" "tools" "utils"No entanto, nem todos são carregados na inicialização do R. Por padrão, apenas os seguintes pacotes são carregados automaticamente:
# [1] "kableExtra" "perm" "coin" "survival" "boot"
# [6] "bootstrap" "future.apply" "future" "plyr" "latticeExtra"
# [11] "lattice" "expm" "Matrix" "knitr" "stats"
# [16] "graphics" "grDevices" "datasets" "utils" "methods"
# [21] "base"Para listar os pacotes carregados, use a função
search()Note que o primeiro elemento, .GlobalEnv, será sempre
carregado pois ele é o ambiente que irá armazenar (e deixar
disponível) os objetos criados pelo usuário. Para carregar um pacote
instalado, usamos a função library(), por exemplo
library(lattice)
search()Isso tornará todas as funções do pacote lattice
disponíveis para uso.
Para instalar um pacote usamos a função
install.packages(). Sabendo o nome do pacote, por exemplo,
mvtnorm, fazemos
install.packages("mvtnorm")Se o diretório padrão de instalação de um pacote for de acesso
restrito (root por exemplo), o R irá perguntar se você gostaria de
instalar o pacote em uma biblioteca pessoal, e sugerirá um diretório que
possui as permissões necessárias. Você pode se antecipar e já definir e
criar um diretório na sua pasta pessoal, e instalar os pacotes sempre
nesse local. Por exemplo, defina ~/R/library como sua
biblioteca pessoal. Para instalar os pacotes sempre nesse diretório
faça:
install.packages("mvtnorm", lib = "~/R/library")Para verificar as bibliotecas disponíveis e se existem pacotes para ser atualizados, use
packageStatus()Para atualizar automaticamente todos os pacotes faça
update.packages(ask = FALSE)3 Criando uma função
A ideia original do R é transformar usuários em programadores
“… to turn ideas into software, quickly and faithfully.”
– John M. Chambers
Criar funções para realizar trabalhos específicos é um dos grandes poderes do R
Por exemplo, podemos criar a famosa função
ola.mundo <- function(){
writeLines("Olá mundo")
}E chama-la através de
ola.mundo()# Olá mundoA função acima não permite alterar o resultado de saída. Podemos fazer isso incluindo um argumento
ola.mundo <- function(texto){
writeLines(texto)
}E fazer por exemplo
ola.mundo("Funções são legais")# Funções são legais(Veremos detalhes de funções mais adiante)
3.1 Exercícios 1
- Usando a função
runif()gere \(30\) números aleatórios entre:- 0 e 1
- -5 e 5
- 10 e 500
alternando a posição dos argumentos da função.
- Veja o help da função (?)
"+" - Crie uma função para fazer a soma de dois números:
xey - Crie uma função para simular a jogada de um dado.
- Crie uma função para simular a jogada de dois dados.
- Crie uma função para simular a jogada de \(n\) dados.
4 Objetos
O que é um objeto?
- Um símbolo ou uma variável capaz de armazenar qualquer valor ou estrutura de dados
Por quê objetos?
- Uma maneira simples de acessar os dados armazenados na memória (o R não permite acesso direto à memória)
Programação:
- Objetos ⇒ Classes ⇒ Métodos
“Tudo no R é um objeto.”
“Todo objeto no R tem uma classe”
- Classe: é a definição de um objeto. Descreve a forma do objeto e como ele será manipulado pelas diferentes funções
- Método: são funções genéricas que
executam suas tarefas de acordo com cada classe. Duas das funções
genéricas mais importantes são:
summary()plot()
Veja o resultado de
methods(summary)
methods(plot)(Veremos mais detalhes adiante).
A variável x recebe o valor \(2\) (tornando-se um objeto dentro do
R):
x <- 2O símbolo <- é chamado de operador de
atribuição. Ele serve para atribuir valores a objetos, e é
formado pelos símbolos < e -,
obrigatoriamente sem espaços.
Para ver o conteúdo do objeto:
x# [1] 2Observação: O símbolo = pode ser usado
no lugar de <- mas não é recomendado.
Quando você faz
x <- 2está fazendo uma declaração, ou seja, declarando que
a variável x irá agora se tornar um objeto que armazena o
número 2. As declarações podem ser feitas uma em cada
linha
x <- 2
y <- 4ou separadas por ;
x <- 2; y <- 4Operações matemáticas em objetos:
x + x# [1] 4Objetos podem armazenar diferentes estruturas de dados:
y <- runif(10)
y# [1] 0.8325578 0.6797345 0.4042401 0.5941045 0.4121373 0.8982319 0.2192148
# [8] 0.8691109 0.7959712 0.3231446Note que cada objeto só pode armazenar uma estrutura (um número ou
uma sequência de valores) de cada vez! (Aqui, o valor \(4\) que estava armazenado em y
foi sobrescrito pelos valores acima.)
4.1 Nomes de objetos
- Podem ser formados por letras, números, “
_”, e “.” - Não podem começar com número e/ou “
_” (começar com ponto não é recomendado) - Não podem conter espaços
- Evite usar acentos
- Evite usar nomes de funções como:
c q t C D F I T diff df data var pt
- O R é case-sensitive, portanto:
dados \(\neq\)
Dados \(\neq\)
DADOS
4.2 Gerenciando a área de trabalho
Liste os objetos criados com a função ls():
ls()Para remover apenas um objeto:
rm(x)Para remover outros objetos:
rm(x, y)Para remover todos os objetos:
rm(list = ls())Cuidado! O comando acima apaga todos os objetos na sua área de trabalho sem perguntar. Depois só é possível recuperar os objetos ao rodar os script novamente.
4.3 Exercícios 2
- Armazene o resultado da equação \(32 +
16^2 - 25^3\) no objeto
x - Divida
xpor \(345\) e armazene emy - Crie um objeto (com o nome que você quiser) para armazenar \(30\) valores aleatórios de uma distribuição uniforme entre \(10\) e \(50\)
- Remova o objeto
y - Remova os demais objetos de uma única vez
- Procure a função utilizada para gerar numeros aleatórios de uma distribuição de Poisson, e gere \(100\) valores para a VA \(X \sim \text{Poisson}(5)\).
5 Tipos e classes de objetos
Para saber como trabalhar com dados no R, é fundamental entender as possíveis estruturas (ou tipos) de dados possíveis. O formato mais básico de dados são os vetores, e a partir deles, outras estruturas mais complexas podem ser construídas. O R possui dois tipos básicos de vetores:
- Vetores atômicos: existem seis tipos básicos:
doubleintegercharacterlogicalcomplexraw
- Listas: também chamadas de vetores recursivos pois listas podem conter outras listas.
A principal diferença entre vetores atômicos e listas é que o primeiro é homogêneo (cada vetor só pode conter um tipo), enquanto que o segundo pode ser heterogêneo (cada vetor pode conter mais de um tipo).
Um vetor atômico só pode conter elementos de um mesmo tipo
Um vetor, como o próprio nome diz, é uma estrutura unidimensional, mas na maioria das vezes iremos trabalhar com estruturas de dados bidimensionais (linhas e colunas). Portanto diferentes estruturas (com diferentes dimensões) podem ser criadas a partir dos vetores atômicos. Quando isso acontece, temos o que é chamado de classe de um objeto. Embora os vetores atômicos só possuam seis tipos básicos, existe um número muito grande de classes, e novas são inventadas todos os dias. E mesmo que um objeto seja de qualquer classe, ele sempre será de um dos seis tipos básicos (ou uma lista).
Para verificar o tipo de um objeto, usamos a função
typeof(), enquanto que a classe é verificada com a função
class(). Vejamos alguns exemplos:
## double
x <- c(2, 4, 6)
typeof(x)# [1] "double"class(x)# [1] "numeric"## integer
x <- c(2L, 4L, 6L)
typeof(x)# [1] "integer"class(x)# [1] "integer"## character
x <- c("a", "b", "c")
typeof(x)# [1] "character"class(x)# [1] "character"## logical
x <- c(TRUE, FALSE, TRUE)
typeof(x)# [1] "logical"class(x)# [1] "logical"## complex
x <- c(2 + 1i, 4 + 1i, 6 + 1i)
typeof(x)# [1] "complex"class(x)# [1] "complex"## raw
x <- raw(3)
typeof(x)# [1] "raw"class(x)# [1] "raw"5.1 Vetores numéricos
Características:
- Coleção ordenada de valores
- Estrutura unidimensional
Usando a função c() para criar vetores:
num <- c(10, 5, 2, 4, 8, 9)
num# [1] 10 5 2 4 8 9typeof(num)# [1] "double"class(num)# [1] "numeric"Por que numeric e não integer?
x <- c(10L, 5L, 2L, 4L, 8L, 9L)
x# [1] 10 5 2 4 8 9typeof(x)# [1] "integer"class(x)# [1] "integer"Para forçar a representação de um número para inteiro é necessário
usar o sufixo L.
Note que a diferença entre numeric e
integer também possui impacto computacional, pois o
armazenamento de números inteiros ocupa menos espaço na memória. Dessa
forma, esperamos que o vetor x acima ocupe menos espaço na
memória do que o vetor num, embora sejam aparentemente
idênticos. Veja:
object.size(num)# 96 bytesobject.size(x)# 80 bytesA diferença pode parecer pequena, mas pode ter um grande impacto computacional quando os vetores são formados por milhares ou milhões de números.
5.1.1 Representação numérica dentro do R
Os números que aparecem na tela do console do R são apenas representações simplificadas do número real armazenado na memória. Por exemplo,
x <- runif(10)
x# [1] 0.2875775 0.7883051 0.4089769 0.8830174 0.9404673 0.0455565 0.5281055
# [8] 0.8924190 0.5514350 0.4566147O objeto x contém números como 0.2875775, 0.7883051,
etc, que possuem 7 casas decimais, que é o padrão do R. O número de
casas decimais é controlado pelo argumento digits da função
options(). Para visualizar essa opção, use
getOption("digits")# [1] 7Note que esse valor de 7 é o número de dígitos significativos, e pode variar conforme a sequência de números. Por exemplo,
y <- runif(10)
y# [1] 0.069360916 0.817775199 0.942621732 0.269381876 0.169348123 0.033895622
# [7] 0.178785004 0.641665366 0.022877743 0.008324827possui valores com 9 casas decimais. Isto é apenas a representação do número que aparece na tela. Internamente, cada número é armazenado com uma precisão de 64 bits. Os únicos números que podem ser representados exatamente no R são os inteiros e frações cujo denominador é potência de 2. Todos os outros números são arredondados internamente com uma acurácia de aproximadamente 53 dígitos binários. Isso pode introduzir algum tipo de erro, por exemplo:
sqrt(2)^2 - 2# [1] 4.440892e-16print(sqrt(2)^2, digits = 22)# [1] 2.000000000000000444089não é exatamente zero, pois a raíz quadrada de 2 não pode ser armazenada com toda precisão com “apenas” 53 dígitos. Outro exemplo:
0.3 + 0.6 - 0.9# [1] -1.110223e-16print(c(0.3, 0.6, 0.9), digits = 22)# [1] 0.2999999999999999888978 0.5999999999999999777955 0.9000000000000000222045Esse tipo de erro é chamado de erro de ponto flutuante, e as operações nessas condições são chamadas de aritmética de ponto flutuante. Para mais informações sobre esse assunto veja What Every Computer Scientist Should Know About Floating-Point Arithmetic e Why doesn’t R think these numbers are equal?.
No R os números podem ser representados com até 22 casas decimais.
Você pode ver o número com toda sua precisão usando a função
print() e especificando o número de casas decimais com o
argumento digits (de 1 a 22)
print(x, digits = 1)# [1] 0.29 0.79 0.41 0.88 0.94 0.05 0.53 0.89 0.55 0.46print(x, digits = 7) # padrão# [1] 0.2875775 0.7883051 0.4089769 0.8830174 0.9404673 0.0455565 0.5281055
# [8] 0.8924190 0.5514350 0.4566147print(x, digits = 22)# [1] 0.28757752012461423873901 0.78830513544380664825439
# [3] 0.40897692181169986724854 0.88301740400493144989014
# [5] 0.94046728429384529590607 0.04555649938993155956268
# [7] 0.52810548804700374603271 0.89241904439404606819153
# [9] 0.55143501446582376956940 0.45661473530344665050507Também é possível alterar a representação na tela para o formato
científico, usando a função format()
format(x, scientific = TRUE)# [1] "2.875775e-01" "7.883051e-01" "4.089769e-01" "8.830174e-01" "9.404673e-01"
# [6] "4.555650e-02" "5.281055e-01" "8.924190e-01" "5.514350e-01" "4.566147e-01"Nessa representação, o valor 2.875775e-01 = \(2.875775 \times 10^{-01}\) = \(0.2875775\).
5.1.2 Sequências de números
Usando a função seq()
seq(1, 10)# [1] 1 2 3 4 5 6 7 8 9 10Ou 1:10 gera o mesmo resultado. Para a sequência variar
em \(2\)
seq(from = 1, to = 10, by = 2)# [1] 1 3 5 7 9Para obter \(15\) valores entre \(1\) e \(10\)
seq(from = 1, to = 10, length.out = 15)# [1] 1.000000 1.642857 2.285714 2.928571 3.571429 4.214286 4.857143
# [8] 5.500000 6.142857 6.785714 7.428571 8.071429 8.714286 9.357143
# [15] 10.000000Usando a função rep()
rep(1, 10)# [1] 1 1 1 1 1 1 1 1 1 1Para gerar um sequência várias vezes
rep(c(1, 2, 3), times = 5)# [1] 1 2 3 1 2 3 1 2 3 1 2 3 1 2 3Para repetir um número da sequência várias vezes
rep(c(1, 2, 3), each = 5)# [1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 35.1.3 Operações matemáticas em vetores numéricos
Operações podem ser feitas entre um vetor e um número:
num * 2# [1] 20 10 4 8 16 18E também entre vetores de mesmo comprimento ou com comprimentos múltiplos:
num * num# [1] 100 25 4 16 64 81num + c(2, 4, 1)# [1] 12 9 3 6 12 105.1.4 A Regra da Reciclagem
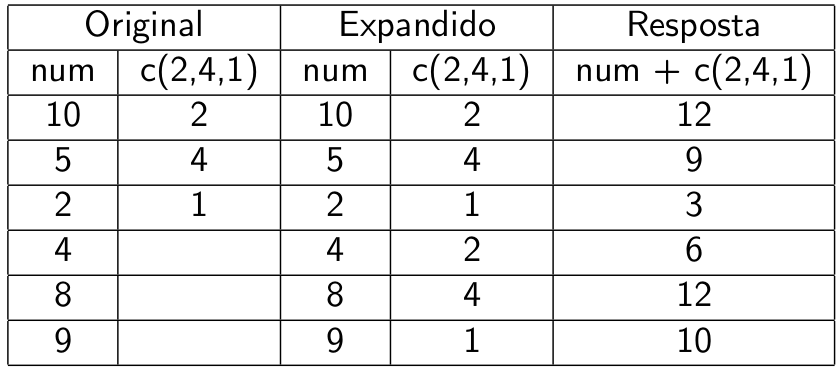
Agora tente:
num + c(2, 4, 1, 3)5.2 Outros tipos de vetores
Vetores também podem ter outros tipos:
- Vetor de caracteres:
caracter <- c("brava", "joaquina", "armação")
caracter# [1] "brava" "joaquina" "armação"typeof(caracter)# [1] "character"class(caracter)# [1] "character"- Vetor lógico:
logico <- caracter == "armação"
logico# [1] FALSE FALSE TRUEtypeof(logico)# [1] "logical"class(logico)# [1] "logical"ou
logico <- num > 4
logico# [1] TRUE TRUE FALSE FALSE TRUE TRUENo exemplo anterior, a condição num > 4 é uma
expressão condicional, e o símbolo > um
operador lógico. Os operadores lógicos utilizados no R
são:
| Operador | Sintaxe | Teste |
|---|---|---|
< |
a < b |
a é menor que b? |
<= |
a <= b |
a é menor ou igual a b? |
> |
a > b |
a é maior que b |
>= |
a >= b |
a é maior ou igual a b? |
== |
a == b |
a é igual a b? |
!= |
a != b |
a é diferente de b? |
%in% |
a %in% c(a, b) |
a está contido no vetor
c(a, b)? |
5.3 Misturando classes de objetos
Algumas vezes isso acontece por acidente, mas também pode acontecer de propósito.
O que acontece aqui?
w <- c(5L, "a")
x <- c(1.7, "a")
y <- c(TRUE, 2)
z <- c("a", T)Lembre-se da regra:
Um vetor só pode conter elementos do mesmo tipo
Quando objetos de diferentes tipos são misturados, ocorre a coerção, para que cada elemento possua a mesma classe.
Nos exemplos acima, nós vemos o efeito da coerção implícita, quando o R tenta representar todos os objetos de uma única forma.
Nós podemos forçar um objeto a mudar de classe, através da
coerção explícita, realizada pelas funções
as.*:
x <- 0:6
typeof(x)# [1] "integer"class(x)# [1] "integer"as.numeric(x)# [1] 0 1 2 3 4 5 6as.logical(x)# [1] FALSE TRUE TRUE TRUE TRUE TRUE TRUEas.character(x)# [1] "0" "1" "2" "3" "4" "5" "6"as.factor(x)# [1] 0 1 2 3 4 5 6
# Levels: 0 1 2 3 4 5 6De ?logical:
Logical vectors are coerced to integer vectors in contexts where a
numerical value is required, with ‘TRUE’ being mapped to ‘1L’,
‘FALSE’ to ‘0L’ and ‘NA’ to ‘NA_integer_’.(x <- c(FALSE, TRUE))# [1] FALSE TRUEclass(x)# [1] "logical"as.numeric(x)# [1] 0 1Algumas vezes não é possível fazer a coerção, então:
x <- c("a", "b", "c")
as.numeric(x)# Warning: NAs introduced by coercion# [1] NA NA NAas.logical(x)# [1] NA NA NA5.4 Valores perdidos e especiais
Valores perdidos devem ser definidos como NA (not
available):
perd <- c(3, 5, NA, 2)
perd# [1] 3 5 NA 2class(perd)# [1] "numeric"Podemos testar a presença de NAs com a função
is.na():
is.na(perd)# [1] FALSE FALSE TRUE FALSEOu:
any(is.na(perd))# [1] TRUEOutros valores especiais são:
NaN(not a number) - exemplo:0/0-InfeInf- exemplo:1/0
A função is.na() também testa a presença de
NaNs:
perd <- c(-1,0,1)/0
perd# [1] -Inf NaN Infis.na(perd)# [1] FALSE TRUE FALSEA função is.infinite() testa se há valores infinitos
is.infinite(perd)# [1] TRUE FALSE TRUE5.5 Exercícios 3
- Crie um objeto com os valores 54, 0, 17, 94, 12.5, 2, 0.9, 15.
- Some o objeto acima com os valores 5, 6.
- Some o o bjeto acima com os valores 5, 6, 7.
- Construa um único objeto com as letras:
A,B, eC, repetidas cada uma 15, 12, e 8 vezes, respectivamente.- Mostre na tela, em forma de verdadeiro ou falso, onde estão as
letras
Bnesse objeto. - Veja a página de ajuda da função
sum()e descubra como fazer para contar o número de letrasBneste vetor (usandosum()).
- Mostre na tela, em forma de verdadeiro ou falso, onde estão as
letras
- Crie um objeto com 100 valores aleatórios de uma distribuição uniforme \(U(0,1)\). Conte quantas vezes aparecem valores maiores ou iguais a 0,5.
- Calcule as 50 primeiras potências de 2, ou seja, \(2, 2^2, 2^3, \ldots, 2^{50}\).
- Calcule o quadrado dos números inteiros de 1 a 50, ou seja, \(1^2, 2^2, 3^2, \ldots, 50^2\).
- Quais pares são iguais, ou seja, quais números inteiros dos dois exercícios anteriores satisfazem a condição \(2^n = n^2\)?
- Quantos pares existem?
- Calcule o seno, coseno e a tangente para os números variando de
\(0\) a \(2\pi\), com distância de \(0.1\) entre eles. (Use as funções
sin(),cos(),tan()).- Calcule a tangente usando a relação \(\tan(x) = \sin(x)/\cos(x)\).
- Calcule as diferenças das tangentes calculadas pela função do R e pela razão acima.
- Quais valores são exatamente iguais?
- Qual a diferença máxima (em módulo) entre eles? Qual é a causa dessa diferença?
6 Outras classes
Como mencionado na seção anterior, o R possui 6 tipos básicos de estrutura de dados, mas diversas classes podem ser construídas a partir destes tipos básicos. Abaixo, veremos algumas das mais importantes.
6.1 Fator
Os fatores são parecidos com caracteres no R, mas são armazenados e tratados de maneira diferente.
Características:
- Coleção de categorias ou níveis (levels)
- Estrutura unidimensional
Utilizando as funções factor() e c():
fator <- factor(c("alta","baixa","baixa","media",
"alta","media","baixa","media","media"))
fator# [1] alta baixa baixa media alta media baixa media media
# Levels: alta baixa mediaclass(fator)# [1] "factor"typeof(fator)# [1] "integer"Note que o objeto é da classe factor, mas seu tipo
básico é integer! Isso significa que cada categoria única é
identificada internamente por um número, e isso faz com que os fatores
possuam uma ordenação, de acordo com as categorias únicas. Por isso
existe a identificação dos Levels (níveis) de um fator.
Veja o que acontece quando “remover a classe” desse objeto
unclass(fator)# [1] 1 2 2 3 1 3 2 3 3
# attr(,"levels")
# [1] "alta" "baixa" "media"Fatores podem ser convertidos para caracteres, e também para números inteiros
as.character(fator)# [1] "alta" "baixa" "baixa" "media" "alta" "media" "baixa" "media" "media"as.integer(fator)# [1] 1 2 2 3 1 3 2 3 3Caso haja uma hierarquia, os níveis dos fatores podem ser ordenados
explicitamente através do argumento levels:
fator <- factor(c("alta","baixa","baixa","media",
"alta","media","baixa","media","media"),
levels = c("alta","media","baixa"))
fator# [1] alta baixa baixa media alta media baixa media media
# Levels: alta media baixatypeof(fator)# [1] "integer"class(fator)# [1] "factor"Além disso, os níveis dos fatores podem também ser explicitamente ordenados
fator <- factor(c("alta","baixa","baixa","media",
"alta","media","baixa","media","media"),
levels = c("baixa", "media", "alta"),
ordered = TRUE)
fator# [1] alta baixa baixa media alta media baixa media media
# Levels: baixa < media < altatypeof(fator)# [1] "integer"class(fator)# [1] "ordered" "factor"(Veja que um objeto pode ter mais de uma classe). Isso geralmente só será útil em casos especificos.
As seguintes funções são úteis para verificar os níveis e o número de níveis de um fator:
levels(fator)# [1] "baixa" "media" "alta"nlevels(fator)# [1] 36.2 Matriz
Matrizes são vetores que podem ser dispostos em duas dimensões.
Características:
- Podem conter apenas um tipo de informação (números, caracteres)
- Estrutura bidimensional
Utilizando a função matrix():
matriz <- matrix(1:12, nrow = 3, ncol = 4)
matriz# [,1] [,2] [,3] [,4]
# [1,] 1 4 7 10
# [2,] 2 5 8 11
# [3,] 3 6 9 12class(matriz)# [1] "matrix" "array"typeof(matriz)# [1] "integer"Alterando a ordem de preenchimento da matriz (por linhas):
matriz <- matrix(1:12, nrow = 3, ncol = 4, byrow = TRUE)
matriz# [,1] [,2] [,3] [,4]
# [1,] 1 2 3 4
# [2,] 5 6 7 8
# [3,] 9 10 11 12Para verificar a dimensão da matriz:
dim(matriz)# [1] 3 4Adicionando colunas com cbind()
cbind(matriz, rep(99, 3))# [,1] [,2] [,3] [,4] [,5]
# [1,] 1 2 3 4 99
# [2,] 5 6 7 8 99
# [3,] 9 10 11 12 99Adicionando linhas com rbind()
rbind(matriz, rep(99, 4))# [,1] [,2] [,3] [,4]
# [1,] 1 2 3 4
# [2,] 5 6 7 8
# [3,] 9 10 11 12
# [4,] 99 99 99 99Matrizes também podem ser criadas a partir de vetores adicionando um atributo de dimensão
m <- 1:10
m# [1] 1 2 3 4 5 6 7 8 9 10class(m)# [1] "integer"dim(m)# NULLdim(m) <- c(2, 5)
m# [,1] [,2] [,3] [,4] [,5]
# [1,] 1 3 5 7 9
# [2,] 2 4 6 8 10class(m)# [1] "matrix" "array"typeof(m)# [1] "integer"6.2.1 Operações matemáticas em matrizes
Matriz multiplicada por um escalar
matriz * 2# [,1] [,2] [,3] [,4]
# [1,] 2 4 6 8
# [2,] 10 12 14 16
# [3,] 18 20 22 24Multiplicação de matrizes (observe as dimensões!)
matriz2 <- matrix(1, nrow = 4, ncol = 3)
matriz %*% matriz2# [,1] [,2] [,3]
# [1,] 10 10 10
# [2,] 26 26 26
# [3,] 42 42 426.3 Array
Um array é a forma mais geral de uma matriz, pois pode ter \(n\) dimensões.
Características:
- Estrutura \(n\)-dimensional
- Assim como as matrizes, podem conter apenas um tipo de informação (números, caracteres)
Para criar um array, usamos a função array(), passando
como primeiro argumento um vetor atômico, e especificamos a dimensão com
o argumento dim. Por exemplo, para criar um objeto com 3
dimensões \(2 \times 2 \times 3\),
fazemos
ar <- array(1:12, dim = c(2, 2, 3))
ar# , , 1
#
# [,1] [,2]
# [1,] 1 3
# [2,] 2 4
#
# , , 2
#
# [,1] [,2]
# [1,] 5 7
# [2,] 6 8
#
# , , 3
#
# [,1] [,2]
# [1,] 9 11
# [2,] 10 12Similarmente, um array de 2 dimensões \(3 \times 2 \times 2\) é obtido com
ar <- array(1:12, dim = c(3, 2, 2))
ar# , , 1
#
# [,1] [,2]
# [1,] 1 4
# [2,] 2 5
# [3,] 3 6
#
# , , 2
#
# [,1] [,2]
# [1,] 7 10
# [2,] 8 11
# [3,] 9 126.4 Lista
Como já vimos, uma lista não é uma “classe” propriamente dita, mas sim um tipo de estrutura de dados básico, ao lado dos vetores atômicos. E, assim como os vetores atômicos, listas são estruturas unidimensionais. A grande diferença é que listas agrupam objetos de diferentes tipos, inclusive outras listas.
Características:
- Pode combinar uma coleção de objetos de diferentes tipos ou classes (é um tipo básico de vetor, assim como os vetores atômicos)
- Estrutura “unidimensional”: apenas o número de elementos na lista é contado
Por exemplo, podemos criar uma lista com uma sequência de números, um caracter e outra lista
lista <- list(1:30, "R", list(TRUE, FALSE))
lista# [[1]]
# [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25
# [26] 26 27 28 29 30
#
# [[2]]
# [1] "R"
#
# [[3]]
# [[3]][[1]]
# [1] TRUE
#
# [[3]][[2]]
# [1] FALSEclass(lista)# [1] "list"typeof(lista)# [1] "list"Para melhor visualizar a estrutura dessa lista (ou de qualquer outro
objeto) poddemos usar a função str()
str(lista)# List of 3
# $ : int [1:30] 1 2 3 4 5 6 7 8 9 10 ...
# $ : chr "R"
# $ :List of 2
# ..$ : logi TRUE
# ..$ : logi FALSENote que de fato é uma estrutura unidimensional
dim(lista)# NULLlength(lista)# [1] 3Listas podem armazenar objetos de diferentes classes e dimensões, por exemplo, usando objetos criados anteriormente
lista <- list(fator, matriz)
lista# [[1]]
# [1] alta baixa baixa media alta media baixa media media
# Levels: baixa < media < alta
#
# [[2]]
# [,1] [,2] [,3] [,4]
# [1,] 1 2 3 4
# [2,] 5 6 7 8
# [3,] 9 10 11 12length(lista)# [1] 26.5 Data frame
Data frame é a versão bidimensional de uma lista. Data frames são listas, mas onde cada componente deve ter obrigatoriamente o mesmo comprimento. Cada vetor da lista vira uma coluna em um data frame, permitindo então que as “colunas” sejam de diferentes tipos.
Os data frames são as estruturas mais comuns para se trabalhar com dados no R.
Características:
- Uma lista de vetores e/ou fatores, de mesmo comprimento
- Pode conter diferentes tipos de dados (numérico, fator, …)
- Estrutura bidimensional
Utilizando a função data.frame():
da <- data.frame(nome = c("João", "José", "Maria"),
sexo = c("M", "M", "F"),
idade = c(32, 34, 30))
da# nome sexo idade
# 1 João M 32
# 2 José M 34
# 3 Maria F 30class(da)# [1] "data.frame"typeof(da)# [1] "list"dim(da)# [1] 3 3Veja os detalhes com str()
str(da)# 'data.frame': 3 obs. of 3 variables:
# $ nome : chr "João" "José" "Maria"
# $ sexo : chr "M" "M" "F"
# $ idade: num 32 34 30Note que a função data.frame() converte caracteres para
fator automaticamente. Para que isso não aconteça, use o argumento
stringsAsFactors = FALSE
da <- data.frame(nome = c("João", "José", "Maria"),
sexo = c("M", "M", "F"),
idade = c(32, 34, 30),
stringsAsFactors = FALSE)
da# nome sexo idade
# 1 João M 32
# 2 José M 34
# 3 Maria F 30str(da)# 'data.frame': 3 obs. of 3 variables:
# $ nome : chr "João" "José" "Maria"
# $ sexo : chr "M" "M" "F"
# $ idade: num 32 34 30Data frames podem ser formados com objetos criados anteriormente, desde que tenham o mesmo comprimento:
length(num)# [1] 6length(fator)# [1] 9db <- data.frame(numerico = c(num, NA, NA, NA),
fator = fator)
db# numerico fator
# 1 10 alta
# 2 5 baixa
# 3 2 baixa
# 4 4 media
# 5 8 alta
# 6 9 media
# 7 NA baixa
# 8 NA media
# 9 NA mediastr(db)# 'data.frame': 9 obs. of 2 variables:
# $ numerico: num 10 5 2 4 8 9 NA NA NA
# $ fator : Ord.factor w/ 3 levels "baixa"<"media"<..: 3 1 1 2 3 2 1 2 2Algumas vezes pode ser necessário converter um data frame para uma matriz. Existem duas opções:
as.matrix(db)# numerico fator
# [1,] "10" "alta"
# [2,] " 5" "baixa"
# [3,] " 2" "baixa"
# [4,] " 4" "media"
# [5,] " 8" "alta"
# [6,] " 9" "media"
# [7,] NA "baixa"
# [8,] NA "media"
# [9,] NA "media"data.matrix(db)# numerico fator
# [1,] 10 3
# [2,] 5 1
# [3,] 2 1
# [4,] 4 2
# [5,] 8 3
# [6,] 9 2
# [7,] NA 1
# [8,] NA 2
# [9,] NA 2Geralmente é o resultado de data.matrix() o que você
está procurando.
Lembre que os níveis de um fator são armazenados internamente como números: \(1^\circ\) nível = 1, \(2^\circ\) nível = 2, \(\ldots\)
fator# [1] alta baixa baixa media alta media baixa media media
# Levels: baixa < media < altastr(fator)# Ord.factor w/ 3 levels "baixa"<"media"<..: 3 1 1 2 3 2 1 2 2as.numeric(fator)# [1] 3 1 1 2 3 2 1 2 27 Atributos de objetos
Um atributo é um pedaço de informação que pode ser “anexado” à qualquer objeto, e não irá interferir nos valores daquele objeto. Os atributos podem ser vistos como “metadados”, alguma descrição associada à um objeto. Os principais atributos são:
namesdimnamesdimclass
Alguns atributos também podem ser visualizados de uma só vez através
da função attributes().
Por exemplo, considere o seguinte vetor
x <- 1:6
attributes(x)# NULLMostra que o objeto x não possui nenhum atributo. Mas
podemos definir nomes, por exemplo, para cada componente desse vetor
names(x)# NULLnames(x) <- c("um", "dois", "tres", "quatro", "cinco", "seis")
names(x)# [1] "um" "dois" "tres" "quatro" "cinco" "seis"attributes(x)# $names
# [1] "um" "dois" "tres" "quatro" "cinco" "seis"Nesse caso específico, o R irá mostrar os nomes acima dos componentes, mas isso não altera como as operaçõs serão realizadas
x# um dois tres quatro cinco seis
# 1 2 3 4 5 6x + 2# um dois tres quatro cinco seis
# 3 4 5 6 7 8Os nomes então podem ser definidos através da função
auxiliar names(), sendo assim, também podemos
remover esse atributo declarando ele como nulo
names(x) <- NULL
attributes(x)# NULLx# [1] 1 2 3 4 5 6Outros atributos também podem ser definidos de maneira similar. Veja os exemplos abaixo:
length(x)# [1] 6## Altera o comprimento (preenche com NA)
length(x) <- 10
x# [1] 1 2 3 4 5 6 NA NA NA NA## Altera a dimensão
length(x) <- 6
dim(x)# NULLdim(x) <- c(3, 2)
x# [,1] [,2]
# [1,] 1 4
# [2,] 2 5
# [3,] 3 6attributes(x)# $dim
# [1] 3 2## Remove dimensão
dim(x) <- NULL
x# [1] 1 2 3 4 5 6Assim como vimos em data frames, listas também podem ter nomes
x <- list(Curitiba = 1, Paraná = 2, Brasil = 3)
x# $Curitiba
# [1] 1
#
# $Paraná
# [1] 2
#
# $Brasil
# [1] 3names(x)# [1] "Curitiba" "Paraná" "Brasil"Podemos também associar nomes às linhas e colunas de uma matriz:
matriz# [,1] [,2] [,3] [,4]
# [1,] 1 2 3 4
# [2,] 5 6 7 8
# [3,] 9 10 11 12attributes(matriz)# $dim
# [1] 3 4rownames(matriz) <- c("A","B","C")
colnames(matriz) <- c("T1","T2","T3","T4")
matriz# T1 T2 T3 T4
# A 1 2 3 4
# B 5 6 7 8
# C 9 10 11 12attributes(matriz)# $dim
# [1] 3 4
#
# $dimnames
# $dimnames[[1]]
# [1] "A" "B" "C"
#
# $dimnames[[2]]
# [1] "T1" "T2" "T3" "T4"Para data frames existe uma função especial para os nomes de linhas,
row.names(). Data frames também não possuem nomes de
colunas, apenas nomes, já que é um caso particular de lista. Então para
verificar/alterar nomes de colunas de um data frame também use
names().
da# nome sexo idade
# 1 João M 32
# 2 José M 34
# 3 Maria F 30attributes(da)# $names
# [1] "nome" "sexo" "idade"
#
# $class
# [1] "data.frame"
#
# $row.names
# [1] 1 2 3names(da)# [1] "nome" "sexo" "idade"row.names(da)# [1] "1" "2" "3"Um resumo das funções para alterar/acessar nomes de linhas e colunas em matrizes e data frames.
| Classe | Nomes de colunas | Nomes de linhas |
|---|---|---|
data.frame |
names() |
row.names() |
matrix |
colnames() |
rownames() |
7.1 Exercícios 4
- Crie um objeto para armazenar a seguinte matriz \[\left[ \begin{array}{ccc} 2 & 8 & 4 \\ 0 & 4 & 1 \\ 9 & 7 & 5 \end{array} \right]\]
- Atribua nomes para as linhas e colunas dessa matriz.
- Crie uma lista (não nomeada) com dois componentes:
(1) um vetor com as letras
A,B, eC, repetidas 2, 5, e 4 vezes respectivamente; e (2) a matriz do exemplo anterior. - Atribua nomes para estes dois componentes da lista.
- Inclua mais um componente nesta lista, com o nome de
fator, e que seja um vetor da classefactor, idêntico ao objetocaractercriado acima (que possui apenas os nomesbrava,joaquina,armação). - Crie um data frame para armazenar duas variáveis: local
(
A,B,C,D), e contagem (42, 34, 59 e 18). - Crie um data frame com as seguintes colunas:
- Nome
- Sobrenome
- Se possui animal de estimação
- Caso possua, dizer o número de animais (caso contrário, colocar 0)
Para criar o data frame, a primeira linha deve ser preenchida com as
suas próprias informações (use a função data.frame()).
Depois, pergunte essas mesmas informações para dois colegas ao seu lado,
e adicione as informações deles à esse data frame (use
rbind()). Acresente mais uma coluna com o nome do time de
futebol de cada um.
8 Programação Orientada a Objetos
Como vimos anteriormente, o R é uma linguagem de programação orientada à objetos. Dois conceitos fundamentais desse tipo de linguagem são os de classe e método. Já vimos também que todo objeto no R possui uma classe (que define sua estrutura) e analisamos algumas delas. O que seria então um método? Para responder essa pergunta precisamos entender inicialmente os tipos de orientação a objetos que o R possui.
O R possui 3 sitemas de orientação a objetos: S3, S4, e RC:
- S3: implementa um estilo de programação orientada a objeto chamada de generic-function. Esse é o estilo mais básico de programação em R (e também o mais utilizado). A ideia é que existam funções genéricas que decidem qual método aplicar de acordo com a classe do objeto. Os métodos são definidos da mesma forma que qualquer função, mas chamados de maneira diferente. É um estilo de programação mais “informal”, mas possibilita uma grande liberdade para o programador.
- S4: é um estilo mais formal, no sentido que que as funções genéricas devem possuir uma classe formal definida. Além disso, é possível também fazer o despacho múltiplo de métodos, ao contrário da classe S3.
- RC: (Reference Classes, antes chamado de R5) é o sistema mais novo implementado no R. A principal diferença com os sistemas S3 e S4 é que métodos pertencem à objetos, não à funções. Isso faz com que objetos da classe RC se comportem mais como objetos da maioria das linguagens de programação, como Python, Java, e C#.
Nesta sessão vamos abordar como funcionam os métodos como definidos pelo sistema S3, por ser o mais utilizado na prática para se criar novas funções no R. Para saber mais sobre os outros métodos, consulte o livro Advanced R.
Vamos entender como uma função genérica pode ser criada através de um
exemplo. Usando a função methods(), podemos verificar quais
métodos estão disponíveis para uma determinada função, por exemplo, para
a função mean():
methods(mean)# [1] mean,ANY-method mean,Matrix-method mean,Raster-method
# [4] mean,sparseMatrix-method mean,sparseVector-method mean,SpatExtent-method
# [7] mean,SpatRaster-method mean,SpatVector-method mean.Date
# [10] mean.default mean.difftime mean.POSIXct
# [13] mean.POSIXlt mean.quosure* mean.units*
# [16] mean.vctrs_vctr* mean.yearmon* mean.yearqtr*
# [19] mean.zoo*
# see '?methods' for accessing help and source codeO resultado são expressões do tipo mean.<classe>,
onde <classe> é uma classe de objeto como aquelas
vistas anteriormente. Isso significa que a função mean(),
quando aplicada à um objeto da classe Date, por exemplo,
pode ter um comportamento diferente quando a mesma função for aplicada à
um objeto de outra classe (numérica).
Suponha que temos o seguinte vetor numérico:
set.seed(1)
vec <- rnorm(100)
class(vec)# [1] "numeric"e queremos calcular sua média. Basta aplicar a função
mean() nesse objeto para obtermos o resultado esperado
mean(vec)# [1] 0.1088874Mas isso só é possível porque existe um método definido
espcificamente para um vetor da classe numeric, que nesse
caso é a função mean.default. A função genérica nesse caso
é a mean(), e a função método é a
mean.default. Veja que não precisamos escrever onome
inteiro da função genérica para que ela seja utilizada, como por
exemplo,
mean.default(vec)# [1] 0.1088874Uma vez passado um objeto para uma função, é a classe do objeto que
irá definir qual método utilizar, de acordo com os métodos disponíveis.
Veja o que acontece se forcarmos o uso da função
mean.Date() nesse vetor
mean.Date(vec)# [1] "1970-01-01"O resultado não faz sentido pois ele é específico para um objeto da
classe Date.
Tudo isso acontece por causa de um mecanismo chamado de
despacho de métodos (method dispatch), que é
responsável por identificar a classe do objeto e utilizar (“despachar”)
a função método correta para aquela classe. Toda função genérica possui
a mesma forma: uma chamada para a função UseMethod(), que
especifica o nome genérico e o objeto a ser despachado. Por exemplo,
veja o código fonte da função mean()
mean# standardGeneric for "mean" defined from package "base"
#
# function (x, ...)
# standardGeneric("mean")
# <environment: 0x55c392678a58>
# Methods may be defined for arguments: x
# Use showMethods(mean) for currently available ones.Agora veja o código fonte da função mean.default, que é
o método específico para vetores numéricos
mean.default# function (x, trim = 0, na.rm = FALSE, ...)
# {
# if (!is.numeric(x) && !is.complex(x) && !is.logical(x)) {
# warning("argument is not numeric or logical: returning NA")
# return(NA_real_)
# }
# if (isTRUE(na.rm))
# x <- x[!is.na(x)]
# if (!is.numeric(trim) || length(trim) != 1L)
# stop("'trim' must be numeric of length one")
# n <- length(x)
# if (trim > 0 && n) {
# if (is.complex(x))
# stop("trimmed means are not defined for complex data")
# if (anyNA(x))
# return(NA_real_)
# if (trim >= 0.5)
# return(stats::median(x, na.rm = FALSE))
# lo <- floor(n * trim) + 1
# hi <- n + 1 - lo
# x <- sort.int(x, partial = unique(c(lo, hi)))[lo:hi]
# }
# .Internal(mean(x))
# }
# <bytecode: 0x55c389d255b8>
# <environment: namespace:base>Agora suponha que você ddeseja criar uma função que calcule a média
para um objeto de uma classe diferente daquelas previamente definidas.
Por exemplo, suponha que você quer que a função mean()
retorne a média das linhas de uma matriz.
set.seed(1)
mat <- matrix(rnorm(50), nrow = 5)
mean(mat)# [1] 0.1004483O resultado é a média de todos os elementos, e não de cada linha. Nesse caso, podemos definir nossa própria função método para fazer o cálculo que precisamos. Por exemplo:
mean.matrix <- function(x, ...) rowMeans(x)Uma função método é sempre definida dessa forma:
<funçãogenérica>.<classe>. Agora podemos ver
novamente os métodos disponíveis para a função mean()
methods(mean)# [1] mean,ANY-method mean,Matrix-method mean,Raster-method
# [4] mean,sparseMatrix-method mean,sparseVector-method mean,SpatExtent-method
# [7] mean,SpatRaster-method mean,SpatVector-method mean.Date
# [10] mean.default mean.difftime mean.matrix
# [13] mean.POSIXct mean.POSIXlt mean.quosure*
# [16] mean.units* mean.vctrs_vctr* mean.yearmon*
# [19] mean.yearqtr* mean.zoo*
# see '?methods' for accessing help and source codee simplesmente aplicar a função genérica mean() à um
objeto da classe matrix para obter o resultado que
desejamos
class(mat)# [1] "matrix" "array"mean(mat)# [1] 0.09544402 0.12852087 0.06229588 -0.01993810 0.23591872Esse exemplo ilustra como é simples criar funções método para
diferentes classes de objetos. Poderíamos fazer o mesmo para objetos das
classes data.frame e list
mean.data.frame <- function(x, ...) sapply(x, mean, ...)
mean.list <- function(x, ...) lapply(x, mean)Aplicando em objetos dessas classes específicas, obtemos:
## Data frame
set.seed(1)
da <- data.frame(c1 = rnorm(10),
c2 = runif(10))
class(da)# [1] "data.frame"mean(da)# c1 c2
# 0.1322028 0.4183230## Lista
set.seed(1)
dl <- list(rnorm(10), runif(50))
class(dl)# [1] "list"mean(dl)# [[1]]
# [1] 0.1322028
#
# [[2]]
# [1] 0.4946632Obviamente esse processo todo é extremamente importante ao se criar
novas funções no R. Podemos tanto criar uma função genérica (como a
mean()) e diversos métodos para ela usando classes de
objetos existentes, quanto (inclusive) criar novas classes e funções
método para elas. Essa é uma das grandes lberdades que o método S3 de
orientação à objetos permite, e possivelmente um dos motivos pelos quais
é relativamente simples criar pacotes inteiros no R.
9 Referências
Para mais detalhes e exemplos dos assuntos abordados aqui, veja Grolemund (2014). Uma abordagem mais avançada e detalhada sobre programação orientada a objetos no R pode ser consultada em (Wickham2015?).
10 Respostas dos exercícios
A resolução dos exercícios desta página está disponível neste script.
11 Informação de Sessão
# Wednesday, 15 February, 2023, 22:10
# ----------------------------------------
# R version 4.2.2 (2022-10-31)
# Platform: x86_64-pc-linux-gnu (64-bit)
# Running under: Ubuntu 22.04.1 LTS
#
# Matrix products: default
# BLAS: /usr/lib/x86_64-linux-gnu/openblas-pthread/libblas.so.3
# LAPACK: /usr/lib/x86_64-linux-gnu/openblas-pthread/libopenblasp-r0.3.20.so
#
# locale:
# [1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
# [4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
# [7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
# [10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
#
# attached base packages:
# [1] stats graphics grDevices datasets utils methods base
#
# other attached packages:
# [1] tidyr_1.2.0 dplyr_1.0.8 kableExtra_1.3.4
# [4] perm_1.0-0.2 coin_1.4-2 survival_3.3-1
# [7] boot_1.3-28 bootstrap_2019.6 future.apply_1.8.1
# [10] future_1.23.0 plyr_1.8.6 latticeExtra_0.6-29
# [13] lattice_0.20-45 expm_0.999-6 Matrix_1.4-1
# [16] knitr_1.37
#
# loaded via a namespace (and not attached):
# [1] matrixStats_0.61.0 sf_1.0-6 webshot_0.5.2
# [4] RColorBrewer_1.1-2 httr_1.4.2 tools_4.2.2
# [7] utf8_1.2.2 R6_2.5.1 KernSmooth_2.23-20
# [10] spData_2.0.1 DBI_1.1.2 colorspace_2.0-2
# [13] raster_3.5-15 sp_1.4-6 tidyselect_1.1.1
# [16] compiler_4.2.2 cli_3.1.1 rvest_1.0.2
# [19] animation_2.7 xml2_1.3.3 microbenchmark_1.4.9
# [22] sandwich_3.0-1 scales_1.1.1 classInt_0.4-3
# [25] mvtnorm_1.1-3 proxy_0.4-26 systemfonts_1.0.3
# [28] stringr_1.4.0 digest_0.6.29 rmarkdown_2.11
# [31] svglite_2.1.0 pkgconfig_2.0.3 jpeg_0.1-9
# [34] htmltools_0.5.2 parallelly_1.30.0 fastmap_1.1.0
# [37] highr_0.9 rlang_1.0.1 rstudioapi_0.13
# [40] generics_0.1.2 shape_1.4.6 jquerylib_0.1.4
# [43] zoo_1.8-9 gtools_3.9.2 spdep_1.2-2
# [46] magrittr_2.0.2 modeltools_0.2-23 s2_1.0.7
# [49] fansi_1.0.2 Rcpp_1.0.8 munsell_0.5.0
# [52] lifecycle_1.0.1 terra_1.5-17 stringi_1.7.6
# [55] multcomp_1.4-18 yaml_2.2.2 MASS_7.3-56
# [58] grid_4.2.2 parallel_4.2.2 listenv_0.8.0
# [61] crayon_1.4.2 deldir_1.0-6 fortunes_1.5-4
# [64] splines_4.2.2 pillar_1.7.0 codetools_0.2-18
# [67] stats4_4.2.2 wk_0.6.0 glue_1.6.1
# [70] evaluate_0.14 renv_0.15.2-7 vctrs_0.3.8
# [73] png_0.1-7 purrr_0.3.4 diagram_1.6.5
# [76] xfun_0.29 libcoin_1.0-9 e1071_1.7-9
# [79] class_7.3-20 viridisLite_0.4.0 tibble_3.1.6
# [82] units_0.8-0 globals_0.14.0 ellipsis_0.3.2
# [85] TH.data_1.1-0Este conteúdo está disponível por meio da Licença Creative Commons 4.0
